Diplomado en Inteligencia Artificial y Aprendizaje Profundo ¶
Clasificación Lineal binaria en Python ¶
Profesores¶
- Alvaro Mauricio Montenegro Díaz, ammontenegrod@unal.edu.co
- Daniel Mauricio Montenegro Reyes, dextronomo@gmail.com
- Campo Elías Pardo Turriago, cepardot@unal.edu.co
Asesora Medios y Marketing digital¶
- Maria del Pilar Montenegro, pmontenegro88@gmail.com
Asistentes¶
- Oleg Jarma, ojarmam@unal.edu.co
- Laura Lizarazo, ljlizarazore@unal.edu.co
Contenido¶
- Introduccion
- Importamos la librerías que usaremos
- Preprocesamiento
- Análisis exploratorio inicial
- Clasificación usando un modelo lineal
- Funciones de pérdida
- Aprendizaje como optimización
- Analizando la función de pérdida cuadrática
- Gradiente descendiente
- Gradiente descendente en batch
- Gradiente descendente estocástico (SGD)
Introducción¶
En esta lección hacemos una primera práctica de modelamiento para clasificación con un subconjunto muy famoso de datos: Iris Dataset.
El propósito del ejercicio es clasificar las flres en dos clases.
El Aprendizaje de Máquina se concentra en la construcción y estudio de sistemas que puedan aprender de los datos. El problema principal consiste en encontrar patrones, relaciones y regularidades sobre los datos, los cuales le permitan construir modelos descriptivos y predictivos. Una aplicación muy común del Aprendizaje de Máquina consiste en la detección de spam, en el cual un modelo recibe un nuevo correo y lo etiqueta como spam o no.
Para construir un modelo de este tipo, se deben tener en cuenta dos conceptos claves. El primero es que el modelo debe debe realizar un proceso automático de clasificación, sin que el usuario le especifique de forma explicita la forma en la que la clasificación se lleve a cabo. Por ejemplo, el modelo recibe ejemplos de correos que son spam y ejemplos de correos que no lo son. El segundo concepto consiste en que el modelo debe tener capidad de generalización, es decir, el modelo debe ser capaz de predecir sobre datos nunca antes vistos. En el ejemplo del filtrado de spam, estamos interesados en clasificar de forma automática los correos que vayan llegando a la bandeja del usuario.
A continuación abordaremos un problema de clasificación binaria sobre un subconjunto del conjunto de datos IRIS.
Originalmente IRIS tiene 150 datos y 4 variables (features). La categorías (target) son 3. Paar los propósistos de esta demostración, se tomaran solamente las especies 1 y 3 y las variables 1 y 2. En consecuencia, el conjunto de datos se reduce a 100 datos, dos variables y dos categorías.
Importamos las librerías que usaremos¶
El siguiente código va a cargar un conjunto de datos (IRIS) que nos va a servir para ilustrar en que consiste un problema de clasificación y como resolverlo con un modelo de machine learning.
#%matplotlib inline
import numpy as np
import matplotlib.pylab as plt
from sklearn import preprocessing
from sklearn import datasets
from io import BytesIO
from PIL import Image
iris = datasets.load_iris()
Preprocesamiento¶
- La variable $y$ contendrá las categorías de las flores. Solamente vamos a conservar las categorias 1 y 2, las cuales recodificamos como -1 y 1.
- La matriz X_noscale contendrá los variables 1 y 2 (ancho de sépalo y ancho de pétalo) y solamente las filas asociadas a las categoría 1 y 2.
- La matriz $X$ es la matriz anterior en donde cada columna está estandarizada de tal manera que tiene media 0 y varianza 1.
y = 2*iris.target[iris.target != 0] - 3
X_noscale = iris.data[:,[1, 3]]
X_noscale = X_noscale[iris.target != 0, :]
X = preprocessing.scale(X_noscale)
Análisis exploratorio inicial¶
Generamos un gráfico con los 100 puntos reservados, los cuales correspondes a dos clases de flores.
def plot_data(X, y):
y_unique = np.unique(y)
colors = plt.cm.rainbow(np.linspace(0.0, 1.0, y_unique.size))
for this_y, color in zip(y_unique, colors):
this_X = X[y == this_y]
plt.scatter(this_X[:, 0], this_X[:, 1], color=color,
alpha=0.5, edgecolor='k',
label="Class: %s" % this_y)
plt.legend(loc="best")
plt.title("Datos")
# dibujamos
plt.figure(figsize=(8, 6))
plt.xlabel('Ancho de sépalo')
plt.ylabel('Ancho de pétalo')
plot_data(X_noscale, y)
Los datos deben ser presentados como arreglos bi-dimensionales de números. Cada fila corresponde a una instancia de entrenamiento, sobre la cual queremos aprender o hacer una predicción sobre sus datos.
Verifiquemos el tamaño de cada arreglo construído
print(X.shape)
print(y.shape)
(100, 2) (100,)
np.unique(y, return_counts=True)
(array([-1, 1]), array([50, 50]))
Observamos que cada clase tiene 50 ejemplos en total. los datos de una clase se representan con una etiqueta positiva, +1, y los de la otra clase con una etiqueta positiva, +1.
print(y)
[-1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1]
Pregunta¶
¿Cómo diferenciamos entre ambas clases de manera automática?
Es decir si me dan un nuevo ejemplo, quiero poderlo clasificar en uno de estas dos clases.
Clasificación usando un modelo lineal¶
Discriminación lineal¶
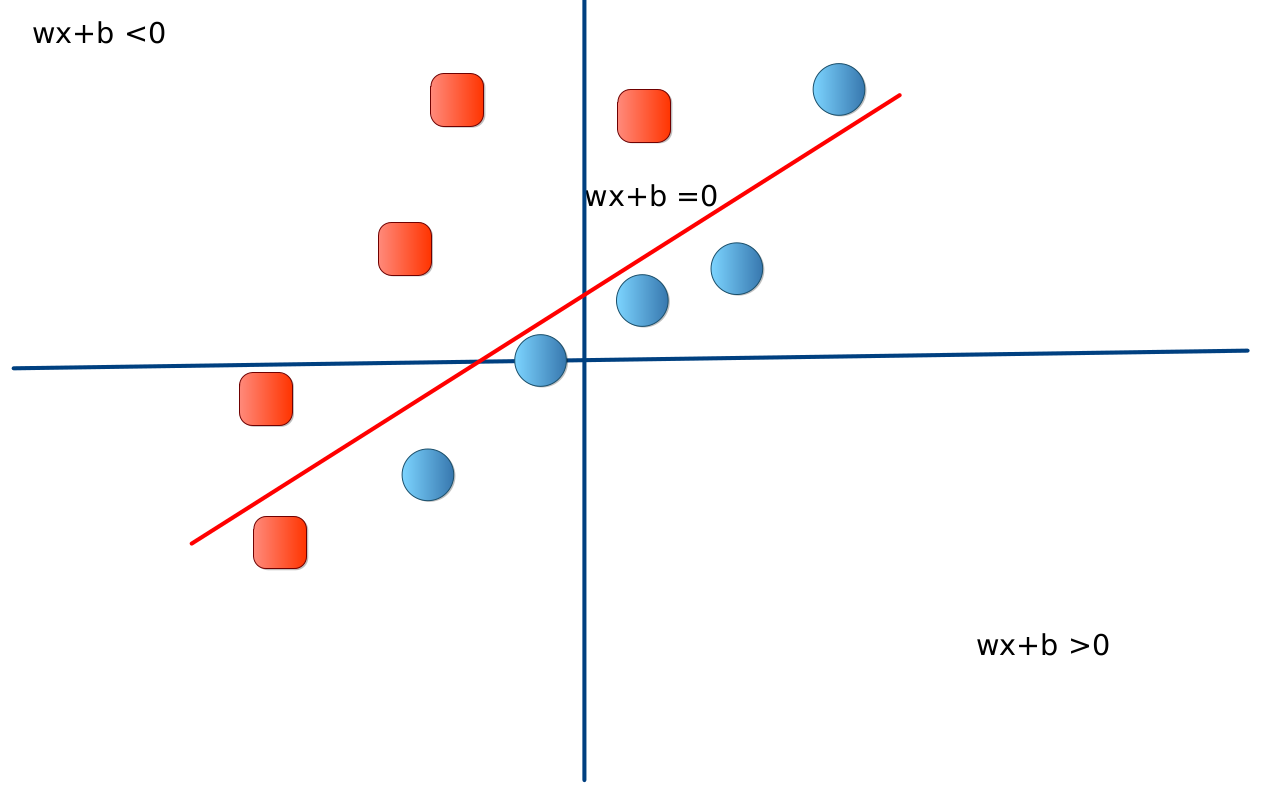
- Nuestro modelo de clasificación es una función que recibe un ejemplo $x$ y retorna la predicción. Esta función se basa en una función (llamada discriminante) $f:\mathbb{R}^{2}\rightarrow\mathbb{R}$ tal que:
- Para el caso de discriminación lineal, definimos $f$ como un modelo lineal con parámetros $w$ y $w_0$:
En la siguiente imagen se ilustra el problema genérico. Se ha tomado $b=w_0$ y $\theta=0$.
Problemas¶
¿Cómo encontrar $f$?
¿Cómo estimamos $w$ y $w_0$?
Funciones de pérdida¶
La pérdida (loss en inglés) es la penalidad por una predicción errada. En otras palabras, la pérdida indica qué tan errada fue la predicción de un modelo en un ejemplo. Si la predicción de mi modelo es perfecta, la pérdida es cero, de lo contrario, la pérdida va a ser un número mayor a cero.
Una función de pérdida muy común es la pérdida cuadrática: $$ L(f, D) =\sum_{(x_{i},r_{i})\in D} (r_i - f(x_i))^2 $$
donde:
- $(x_{i},r_{i})$ es un ejemplo en el cual $x_i$ corresponde a un conjunto de características, en nuestro conjunto de datos, corresponde a las 2 características extraídas de una flor (Ancho del sépalo y ancho del pétalo). Estas características son usadas por el modelo para hacer predicciones. $r_i$ corresponde a la etiqueta del ejemplo, por ejemplo la especie de la flor.
- $f(x_i)$ corresponde a la función de predicción que definimos previamente. Esta función es de la forma $f(x) = wx+w_0$.
- $D$ corresponde al conjunto de datos compuesto por varios ejemplos anotados.
Aprendizaje como optimización¶
- Estimar los parámetros $w$ y $w_0$ puede ser abordado como un problema de optimización que consiste en:
dónde $L(f, D)$ es la función de pérdida cuadrática. Esto se resume en encontrar una función $f$ que genere el valor mínimo de pérdida promedio con respecto a todos los ejemplos del conjunto de datos.
- La función $f$ proviene de una conjunto de funciones llamado el espacio de hipótesis:
dónde $w$ y $w_0$ son los coeficientes de la función $f(x) = wx+w_0$.
- La función de pérdida nos ayuda a estimar qué tan mal se comporta una función $f$ del espacio de hipótesis con respecto al conjunto de datos $D$.
A continuación, definimos en Python las funciones de predicción y pérdida, para nuestro problema.
def predict(w, x):
a = np.dot(w[1:], x) + w[0]
return a
def square_loss(w, x, y):
return (y - predict(w, x)) ** 2 / 2
def batch_loss(loss_fun, w, X, Y):
n = X.shape[0]
tot_loss = 0
for i in range(n):
tot_loss += loss_fun(w, X[i], Y[i])
return tot_loss
Supongamos $w$ y $w_0$:
- $w = [5, 2]$
- $w_0 = 1$
Escogemos el primer ejemplo de nuestro conjunto de datos Iris:
w = [1, 5, 2] # Por facilidad, w[0] es igual a w_0
x = X[0] # Primer ejemplo de nuestro conjunto de datos IRIS.
label = y[0]
print('Características: {}'.format(X[0]))
print('Etiqueta real: {}'.format(y[0]))
print('Función discriminante: {}'.format(predict(w, x)))
Características: [ 0.99068792 -0.65303909] Etiqueta real: -1 Función discriminante: 4.647361438018154
Puesto que el valor de la función discriminante es $f(x)>0$, la clase predicha es positiva. Ahora, estimamos el valor de pérdida usando esos valores de $w$ y $w_0$
print('Pérdida: {}'.format(square_loss(w, x, label)))
Pérdida: 15.946345605807238
¿Qué pasa si modificamos uno de los parámetros $w$?
w_p = [1, 5, 3]
print('Etiqueta: {}'.format(y[0]))
print('Predicción: {}'.format(predict(w_p, x)))
print('Pérdida w_p: {}'.format(square_loss(w_p, x, label)))
Etiqueta: -1 Predicción: 3.9943223502437437 Pérdida w_p: 12.471627869072096
Observamos que nuestra pérdida bajó de $15.94$ a $12.47$. Sin embargo, estamos interesados en automatizar este proceso. Primero vamos a observar cómo se comporta la función de pérdida cuadrática.
Analizando la función de pérdida cuadrática¶
A continuación, definimos un par de funciones de python para poder visualizar la función de pérdida
def plot_loss(loss):
w1_vals = np.linspace(-5, 5, 30)
w2_vals = np.linspace(-5, 5, 30)
W1, W2 = np.meshgrid(w1_vals, w2_vals)
grid_r, grid_c = W1.shape
ZZ = np.zeros((grid_r, grid_c))
for i in range(grid_r):
for j in range(grid_c):
ZZ[i, j] = loss(W1[i, j], W2[i, j])
pl.contourf(W1, W2, ZZ,30, cmap = pl.cm.jet)
pl.colorbar()
pl.xlabel("$w_1$")
pl.ylabel("$w_2$")
def bloss_square(w1, w2):
w = np.array([1, w1, w2])
return batch_loss(square_loss, w, X, y)
plot_loss(bloss_square)
Intepretación: Observamos que el valor mínimo de nuestra función de pérdida se encuentra alrededor de:
- $w_1 = 0.0$
- $w_2 = 1.0$
Gradiente descendiente¶
- Ventajas en este caso:
- Óptimo global garantizado
- Simplicidad del método
- Facilidad para ajustar los parámetros
- Escalabilidad
- Paralelización potencial.
- En aprendizaje de máquina, las preferencias cambian sobre el tiempo.
- Hoy en día es escalable. Inclusive, la estrategias paralelizables son preferidas a sabiendas de la optimalidad garantizada.
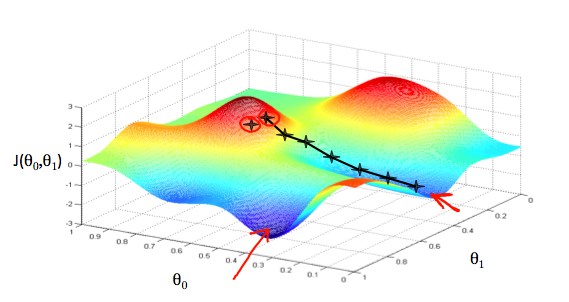
Minimizar la función $L(f, D)$, donde $f$ es nuestra función de predicción resulta ser un problema convexo, es decir, existe solo un lugar donde la inclinación de la curva de pérdida es igual a cero. Ese valor mínimo es dónde nuestra función de pérdida $L$ converge.
Por otro lado, calcular el valor de la función de pérdida para cada posible valor de $w$ sobre el conjunto de datos es una forma muy ineficiente de hallar ese valor de convergencia. Gradiente descendente es un mecanismo iterativo que nos permite hayar ese valor de convergencia.
Algoritmo gradiente descendente:¶
- Paso 1: Escogemos un valor inicial o punto de partida. Puede ser cero o un valor aleatorio. Este valor inicial no tiene relevancia para este problema.
- Paso 2: Calculamos el gradiente de la función de pérdida para el punto de partida. El gradiente de la pérdida me indica la derivada (inclinación) de la curva. El gradiente a su vez es un vector, por lo tanto tiene dirección y magnitud, esta dirección siempre apunta hacía donde se genera el mayor incremento en la función de pérdida.
- Paso 3: Calculamos un nuevo valor $w$ cambiándolo en la dirección negativa del gradiente, con el objetivo reducir la pérdida lo más pronto posible.
- Paso 4: El nuevo valor de $w$ también estará determinado por la magnitud del gradiente y una taza de aprendizaje $\eta$. Por ejemplo, si la magnitud del gradiente es 2.5 y la taza de aprendizaje es 0.01, gradiente descendente escogerá un punto $w$ que esté alejado 0.025 unidades del punto anterior $w$. Para el calculo de $w$ usaremos la siguiente formula:
- Repetir desde el Paso 1.
Problemas¶
- ¿Qué pasa si el tamaño de nuestro paso es muy grande y no alcanza el minimo local?
La elección de una tasa de aprendizaje alta puede terminar en que nunca alcanzemos el punto de convergencia. Pero una tasa muy pequeña puede llevar a que tome mucho tiempo llegar al mínimo global
¿Cómo se calcula el gradiente?¶
Si $f_w$ es la función que definimos antes se tiene que
$$ \frac{\partial E(w,x_{i,}r_{i})}{\partial w_1}=(w_0 + w_1x_i-r_{i})x_{i} $$y $$ \frac{\partial E(w,x_{i,}r_{i})}{\partial w_0}=(w_0 + w_1x_i-r_{i}) $$
A continuación, definimos una función de_dw que corresponde el derivadas parciales de $E$ con respecto a los coeficientes $w$.
def de_dw(w, x, r):
x_prime = np.zeros(len(x) + 1)
x_prime[1:] = x
x_prime[0] = 1
return (predict(w, x) - r) * x_prime
Gradiente descendente en batch¶
Para estimar el gradiente, lo hacemos a lo largo de todos nuestro conjunto de datos. Puesto que solo contamos con 100 ejemplos, es relativamente rápido hacer este calculo. Sin embargo, en la práctica puede ser ineficiente si se cuenta con un gran número de ejemplos de entrenamiento.
def batch_gd(X, Y, epochs, eta, w_ini):
"""
X: instancias del conjunto de datos
Y: etiquetas del conjunto de datos
epochs: número de iteraciones para ejecutar
gradiente descendente
eta: taza de aprendizaje
w_ini: w y w_0 iniciales
"""
losses = []
w = w_ini
n = X.shape[0]
for i in range(epochs):
delta = np.zeros(len(w))
for j in range(n):
delta += de_dw(w, X[j], Y[j]) # Vamos sumando el gradiente por cada ejemplo en el conjunto de datos
w = w - eta * delta # Calculamos el nuevo valor de w
losses.append(batch_loss(square_loss, w, X, Y)) # Vamos guardando el valor de pérdida para visualizar luego
return w, losses
Vamos a ejecutar gradiente descendente en batch con los siguientes parámetros:
- $epochs = 50$
- $w_0 = 0$ ($w_0$ inicial)
- $w = [0, 0]$ ($w$ inicial)
- $\eta = 0.01$ (tasa de aprendizaje)
w, losses = batch_gd(X, y, 50, 0.001, np.array([0, 0, 0]))
pl.figure(figsize = (8,16/3))
pl.plot(losses)
plt.show()
Gradiente descendente estocástico (SGD)¶
Cuando trabajamos con grandes conjuntos de datos, se vuelve poco práctico el cálculo del gradiente sobre todo el conjunto de datos. Una forma de disminuir el tiempo de computación es escogiendo muestras al azar de nuestro conjunto de datos, que nos generan un estimado en promedio del gradiente promedio. SGD toma entonces una muestra de forma aleatoria a la vez y estima el valor del gradiente para esa muestra. A pesar de ser ruidoso, SGD funciona bastante bien en la práctica.
def sgd(X, Y, epochs, eta, w_ini):
"""
X: instancias del conjunto de datos
Y: etiquetas del conjunto de datos
epochs: número de iteraciones para ejecutar
gradiente descendente
eta: tasa de aprendizaje
w_ini: w y w_0 iniciales
"""
losses = []
w = w_ini
n = X.shape[0]
index = list(range(n))
for i in range(epochs):
np.random.shuffle(index)
for j in index:
delta = de_dw(w, X[j], Y[j]) # Aquí estimamos el gradiente para cada elemento pero no para todo el dataset
w = w - eta * delta
losses.append(batch_loss(square_loss, w, X, Y))
return w, losses
Ejemplo de orden aleatorio
index = list(range(10))
print(index)
np.random.shuffle(index)
print(index)
[0, 1, 2, 3, 4, 5, 6, 7, 8, 9] [5, 1, 7, 2, 9, 8, 4, 6, 0, 3]
A continuación, comparamos el comportamiento de Gradiente Descendente en Batch y Estocástico. Observamos que tienen un comportamiento similar para nuestro problema de clasificación de dos clases.
lr = 0.005
epochs = 500
w1, losses_bt = batch_gd(X, y, epochs, lr, np.array([0, 0, 0])) #Batch
w2, losses_ol = sgd(X, y, epochs, lr, np.array([0, 0, 0])) #SGD
pl.figure(figsize = (8,16/3))
pl.plot(np.arange(epochs), losses_ol, label="SGD")
pl.plot(np.arange(epochs), losses_bt, label="Batch")
pl.xlabel("Epoch")
pl.ylabel("Loss")
pl.legend()
plt.show()
Visualización de la función de predicción¶
Es útil poder visualizar qué regiones de nuestro espacio en 2D son asignadas a la clase positiva y a la clase negativa. Para eso escribimos una función que nos permite visualizar:
def plot_decision_region(X, pred_fun):
"""
X: corresponde a las instancias del conjunto de datos
pred_fun: es una función que para cada valor de X, regresa una predicción
"""
min_x = np.min(X[:, 0])
max_x = np.max(X[:, 0])
min_y = np.min(X[:, 1])
max_y = np.max(X[:, 1])
min_x = min_x - (max_x - min_x) * 0.05
max_x = max_x + (max_x - min_x) * 0.05
min_y = min_y - (max_y - min_y) * 0.05
max_y = max_y + (max_y - min_y) * 0.05
x_vals = np.linspace(min_x, max_x, 30)
y_vals = np.linspace(min_y, max_y, 30)
XX, YY = np.meshgrid(x_vals, y_vals)
grid_r, grid_c = XX.shape
ZZ = np.zeros((grid_r, grid_c))
for i in range(grid_r):
for j in range(grid_c):
ZZ[i, j] = pred_fun(XX[i, j], YY[i, j])
pl.contourf(XX, YY, ZZ, 30, cmap = pl.cm.coolwarm, vmin= -1, vmax=2)
pl.colorbar()
pl.xlabel("x")
pl.ylabel("y")
def gen_pred_fun(w):
def pred_fun(x1, x2):
x = np.array([x1, x2])
return predict(w, x)
return pred_fun
Visualizemos el caso donde nuestros coeficiente sean de la forma:
- $w_0 = 0$
- $w = [0.6, 1]$
w = [0, 0.6, 1]
pl.figure(figsize = (8,16/3))
plot_decision_region(X, gen_pred_fun(w))
plot_data(X, y)
Nada útil, sin embargo si visualizamos el resultado luego de aplicar SGD:
lr = 0.005
epochs = 500
w, _ = sgd(X, y, epochs, lr, np.array([0, -1, 2])) #SGD
pl.figure(figsize = (8,16/3))
plot_decision_region(X, gen_pred_fun(w))
plot_data(X, y)
w
array([ 0.03780429, -0.23722539, 0.94336809])